Ich möchte eine Clustermap / Heatmap von Daten zur Anwesenheit / Abwesenheit von Genen von Patienten erstellen, bei denen die Gene in Kategorien (z. B. Chemotaxis, Endotoxin usw.) eingeteilt und entsprechend gekennzeichnet werden. Ich habe keine solche Option in der Dokumentation von Seaborn gefunden. Ich weiß, wie man die Heatmap generiert, ich weiß nur nicht, wie man Yticks als Kategorien kennzeichnet. Hier ist ein Beispiel (unabhängig von meiner Arbeit) von dem, was ich erreichen möchte:
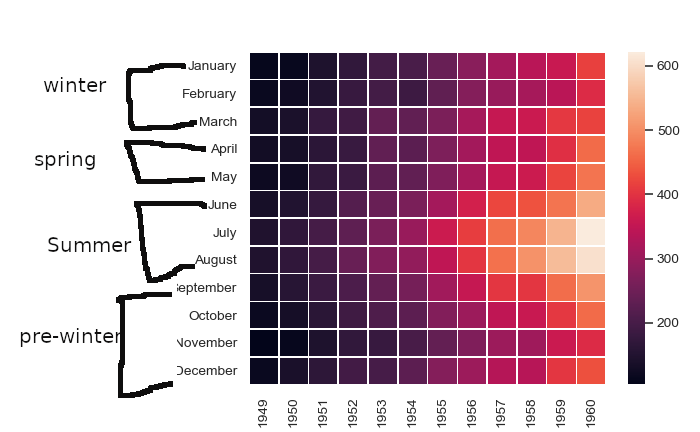
Hier erhalten die Yticklabels Januar, Februar und März das Gruppenlabel Winter und andere Yticklabels sind ebenfalls ähnlich gekennzeichnet.

Antworten:
Ich habe das Beispiel, das Sie in seaborn gegeben haben, reproduziert und die Antwort von @ Stein von hier aus angepasst .
Gibt:
Ich hoffe, das hilft.
quelle
Ich habe dies noch nicht mit Seaborn getestet, aber das Folgende funktioniert mit Vanille-Matplotlib.
quelle